유전자 마이크로어레이는 대표적인 생명정보 대량 획득기술의 하나로 생명정보학 (bioinformatics)의 발전에 크게 기여하였다.
하나의 칩 위에서 수만 개의 유전자 발현을 정량적으로 측정하는 것이므로 복잡한 생물체를 이해하는 새로운 접근을 가능하게 하였다.
그러나 마이크로어레이 실험으로 생성되는 데이터는 형태가 다양하고 방대하기 때문에 전산적인 도구 없이는 데이터를 분석하고 관리하기 어렵다.
유전자 발현 데이터는 개개 유전자 및 조직표본에 대한 정보와 실험조건, 실험방법들에 대한 상세한 주석이 있어야 해석이 가능하기 때문에 다차원적인 자료구조를 필요로 한다. 이런 데이터의 복잡성 때문에 데이터를 체계적으로 관리하고 연구자들 간에 데이터를 공유하기에 어려움이 있다.
MGED (Microarray Gene Expression Data) 단체에서는 마이크로어레이 데이터를 공유할 수 있도록 표준화 작업을 진행해왔다.
MIAME (Minimal Information About a Microarray Experiment)는 마이크로어레이 실험을 보고할 때 필요한 최소한의 내용을 정의한 것이다.
MAGE-OM (Microarray Gene Expression Object Model)은 마이크로어레이의 표준 객체 모델이고, MAGE-ML (Microarray Gene Expression Markup Language)은 MAGE-OM을 XML (Extensible Markup Language) 형식으로 구현한 표준 데이터교환 포맷이다.
MGED의 표준화 제안이 공식적으로 인정받게 되면서, 표준을 지원하는 마이크로어레이 데이터베이스 개발 노력이 증가하고 있다. 그러나 단순한 MIAME 데이터 요구를 반영하는 데이터베이스가 대부분이고 표준 데이터 모델이나 형식을 완벽하게 지원해주는 데이터베이스의 개발은 미비한 상황이다.
|
마이크로어레이 데이터를 체계적으로 관리하고 외부 시스템과 호환이 가능한 마이크로어레이 정보시스템 (Microarray Information System)을 계획하였다.
MGED 표준을 따르는 정보 시스템은 두 개의 하위 시스템으로 구성된다.
하나는 MIAME 표준을 적용한 데이터베이스 (MIAME 데이터베이스)가 중심이 되는 연구실 정보 관리 시스템 (Laboratory Information Management System: LIMS)이다. MIAME 데이터 요구에 따라 데이터를 저장하고, MAGE-ML 표준 형식으로 데이터를 출력하여 다른 데이터베이스와 교류가 가능하다.
다른 하나는 표준 데이터 모델인 MAGE-OM을 구현한 데이터베이스 (MAGE-OM 데이터베이스)가 중심이 되는 시스템이다.
MAGE-OM 데이터베이스는 다른 출처로부터 모아진 MAGE-ML을 저장하여 종합적인 분석을 할 수 있는 중앙 데이터베이스이다. 객체지향 모델의 풍부한 표현력을 활용할 수 있고, 표준 모델을 그대로 구현한 것이므로 유전자 발현 데이터를 외부 자원과 연결할 수 있는 기반이 된다.
마이크로어레이 정보 시스템은 유전자 발현 데이터를 필요로 하는 생물학자, 정보학자 간의 데이터 연결을 용이하게 하며, 표준 데이터 형식인 MAGE-ML 문서가 입출력 되므로 외부 시스템과 호환이 가능하다. |
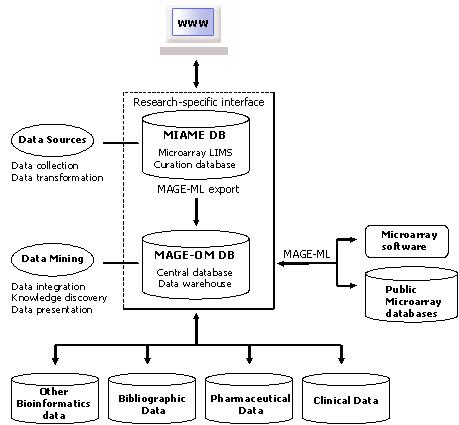 |
MAGE-ML 변환 시스템은 MIAME 데이터베이스의 관계형 데이터를 XML 기반의 MAGE-ML로 변환하여 공식적인 데이터 교류를 가능하게 한다.
MAGE-ML 문서는 관계형 테이블에 분산되어 있는 정보를 연속된 형태로 나타나므로 하나의 실험에 대한 정보를 효율적으로 처리할 수 있다는 장점이 있다.
|
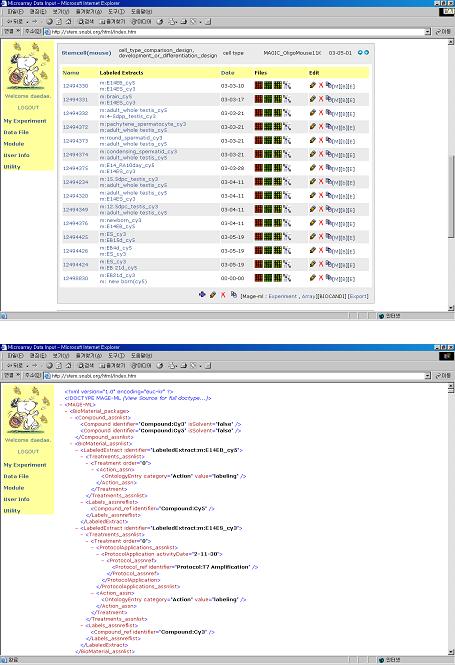
MAGE-ML에서 유전자 발현 데이터의 표현 |
| |
back to main page |


 연구배경
연구배경 연구계획
연구계획
 MAGE-ML 변환 프로그램
MAGE-ML 변환 프로그램